Coronavirus y divagaciones genéticas
lunes , 25 de mayo de 2020

Coronavirus y divagaciones genéticas
Parafraseando el título de una famosa película protagonizada por Charlton Heston (‘El planeta de los simios’), hago mías las palabras del periodista Juan Ochoa de Eribe (El Mundo, 8 abril 2020, p.18) que decía que habitamos en el “planeta de los virus”. Se calcula que existen más de 100 millones de tipos diferentes de virus, infectando a 1.750.000 especies distintas, jugando un papel importante en nuestro ecosistema. La inmensa mayoría son inocuos para la humanidad. Hasta 2012 solamente se habían identificado 219 tipos (¿especies?) de virus capaces de infectar al ser humano, descubriéndose 3 ó 4 nuevos tipos cada año. Al menos dos tercios serían capaces de infectar también otros hospedadores no humanos, principalmente mamíferos y, en ocasiones, aves. Unos 110 tipos de virus son capaces de transmitirse entre personas y solamente 55 tienen capacidad epidémica. En los océanos existen diez mil millones de trillones (1031) de partículas víricas (Venter, 2007, 344).
Seguir leyendo
Aunque parezca algo apocalíptico, las citas que incluyo a continuación sobre el ADN egoísta tienen que hacernos reflexionar. ¿Qué sentido biológico tiene una pandemia como la del SARS CoV-2? ¿Y desde el punto de vista evolutivo?
– “El ADN ni se preocupa ni conoce. El ADN nada más es. Y nosotros danzamos al son de su música” (Richard Dawkins).
– “El ADN proporciona la música. Nuestras células y el ambiente proporcionan la orquesta” (J. Craig Venter).
– “No hay música sin instrumentos, nosotros somos los instrumentos”.
– “Los virus son malas noticias envueltas en proteínas” (Peter Medawar, Premio Nobel en Fisiología o Medicina 1960). Las malas noticias pueden venir escritas en dos alfabetos distintos, según que su genoma sea de ADN o de ARN (Coronavirus).
– En esta pandemia yo estoy viendo al ARN y al ADN como máquinas monstruosas que se alimentan a sí mismas matando las células y al organismo humano.
Hay varias formas de clasificar los virus, pero desde una perspectiva genética me parece más adecuado hacer una clasificación en función de su organización genética: material hereditario o genoma (ADN o ARN), tipo de molécula (circular o lineal), tipo de hélice (sencilla o doble) y, finalmente, tipo de hospedador al que parasita (bacteria, animal o vegetal), tal como propuso David Baltimore, Premio Nobel en Fisiología o Medicina 1975 “por sus descubrimientos en relación con la interacción entre los virus tumorales y el material genético de la célula”. Al final, lo que interesa es cómo se llega al ARN mensajero (ARNm) en la célula para producir la síntesis de las proteínas víricas que permitan la reproducción del virus.
Coronavirus
Los coronavirus son virus con genoma ARN lineal de hélice sencilla de tipo positivo (ARN+) que en su ciclo reproductivo, por tanto, debe pasar a ARN de tipo negativo (ARN-) para dar lugar al ARN mensajero (ARNm) codificador de las proteínas víricas.
El coronavirus (SARS CoV-2) causante de la enfermedad COVID-19 pandémica actual está relacionado con otros coronavirus responsables de epidemias pasadas como son el MERS CoV que produjo el Síndrome Respiratorio de Oriente Medio (Arabia Saudí, 2002; tasa de mortalidad del 30%) y le SARS CoV del Síndrome Respiratorio Agudo y Grave de China (2002; tasa de mortalidad del 10%). Indico a continuación alguna de sus características:
– 50 / 200 nm de diámetro.
– Material genético: ARN monocatenario (mc) lineal de unas 30.000 bases nitrogenadas. ARN mc (+) → ARN mc (-) → ARNm.
– Cápside: envoltura proteica que puede estar rodeada por una membrana.
– Cuatro genes codificantes para proteínas estructurales:
S: Homotrímero de glicoproteína (forman las ‘espículas’ que sobresalen y son reconocidas por los receptores de las células).
E: Proteína pequeña de envoltura.
M: Proteína de la matriz que une la envoltura al núcleo vírico.
N: Fosfoproteína de la nucleocápside.
– ORFs: Proteínas no estructurales, incluyendo las enzimas que aparecen durante su ciclo reproductivo dentro del hospedador.
¿Cuántas nuevas partículas víricas produce cada virus?
Coronavirus y Genética de Poblaciones
La evolución de los seres vivos se produce gracias a la variabilidad genética de las poblaciones y a que las circunstancias ambientales físicas o biológicas (mutaciones favorables o desfavorables, agentes patógenos) actúan sobre los organismos según sea su genotipo, favoreciéndoles o perjudicándoles (selección natural darwiniana), pero debe quedar bien claro el carácter preadaptativo de la mutación: esto es, la mutación existe en la población antes de que se presenten los factores físicos o biológicos que puedan afectar a la viabilidad de los individuos. Hay un ejemplo típico para explicarlo: Suele decirse, equivocadamente, que los antibióticos producen en las bacterias mutaciones que las hacen resistentes a los mismos, haciendo ineficaz el posterior tratamiento con dicho antibiótico. Lo que realmente sucede es que en la población de bacterias existen genotipos distintos que las hacen susceptibles o resistentes al antibiótico antes de que este haya estado en contacto con las bacterias. Al hacer el tratamiento muere la mayoría de las bacterias porque son susceptibles al antibiótico, pero sobreviven unas pocas que son resistentes y, entonces, al reproducirse dan lugar a una nueva población bacteriana resistente al antibiótico.
En resumen, la mutación existe antes de que sea útil a los individuos que la poseen: carácter preadaptativo de la mutación, frente al carácter postadaptativo que significaría que la mutación surge como respuesta al antibiótico.
Al escribir estas líneas me viene a la mente la insensatez del Premier británico, Boris Johnson, cuando propuso que se dejara infectar con el coronavirus a toda la población británica para que sobrevivieran las personas genéticamente resistentes y así quedara un Reino Unido inmune a la infección.
Genes, pueblos y lenguas
Para entender mejor lo que está ocurriendo con la pandemia del coronavirus, puede ser útil hacer referencia a los trabajos del profesor Luigi Luca Cavalli-Sforza (1991) sobre “genes, pueblos y lenguas”. El hecho probado por Cavalli-Sforza y colaboradores (1988) es que hay un paralelismo entre la evolución genética humana y las lenguas, de manera que se puede estimar que existen unas 5.000 poblaciones (¿etnias?) diferentes que se corresponden con las aproximadamente 5.000 lenguas habladas en la actualidad, clasificadas en 17 familias lingüísticas. Los árboles genealógicos que relacionan pueblos y lenguas se superponen.
En otro estudio posterior (Sutherland, 2003), se estimó que en el mundo se hablan unas 6.800 lenguas de las que entre 900 y 1.000 están en peligro de extinción y más de 700 en estado vulnerable, aunque las causas de extinción sean distintas.
En este contexto me viene a la memoria la idea que expone el Premio Nobel de Literatura José Saramago en su obra ‘La caverna’ (Alfaguara, 2000) en la que describe cómo una familia de alfareros comprende que ha dejado de ser útil al mundo. Dice así:
“Todos los días se extinguen especies animales y vegetales, todos los días hay profesiones que se vuelven inútiles, idiomas que dejan de tener personas que los hablen, tradiciones que pierden sentido, sentimientos que se convierten en sus contrarios”.
¿Y qué tiene que ver todo esto con el coronavirus? ¿por qué mezclo ‘churras con merinas’? La explicación es la siguiente: el profesor Ángel Gómez Moreno, Catedrático de Literatura Española de la Universidad Complutense ha escrito un artículo (Gómez Moreno, 2020) sobre la distribución de la pandemia de la COVID-19 producida por el coronavirus SARS CoV-2. En su artículo, el profesor Gómez Moreno recoge los datos de la pandemia del coronavirus por Europa en relación con la frecuencia con que aparece el haplotipo R1b del ADN-Y (cromosoma Y) en los varones de las distintas poblaciones (gráficos 1, 2 y 3).
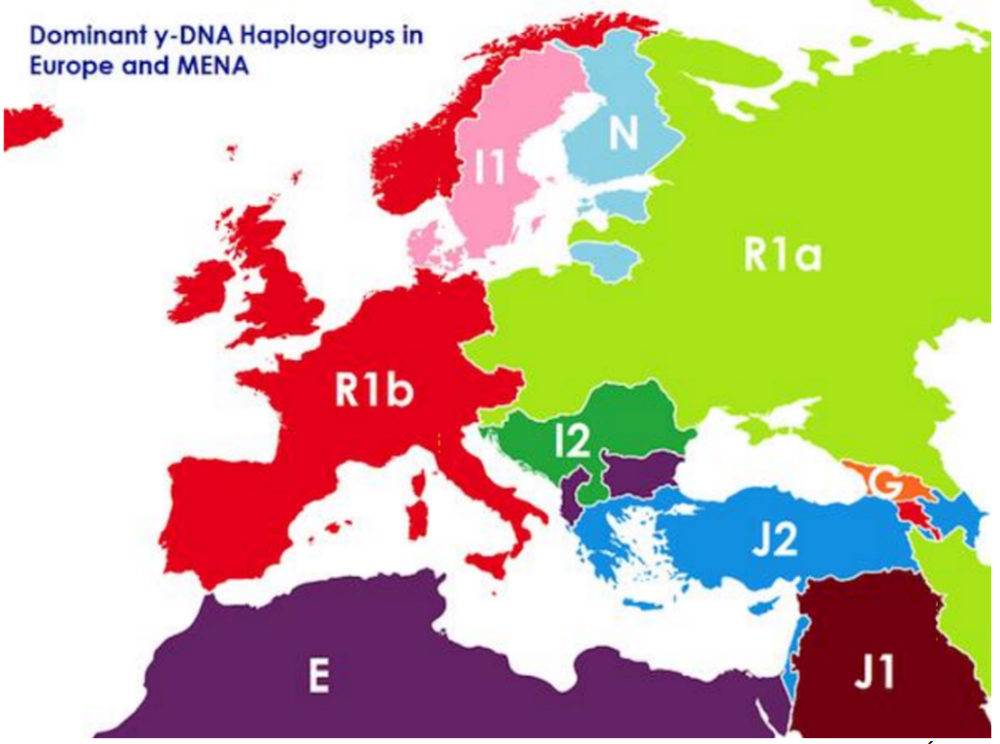
Gráfico 1. Mapa de los haplogrupos predominantes en Europa y zonas próximas de África y Asia (Gómez Moreno, 2020, 25)
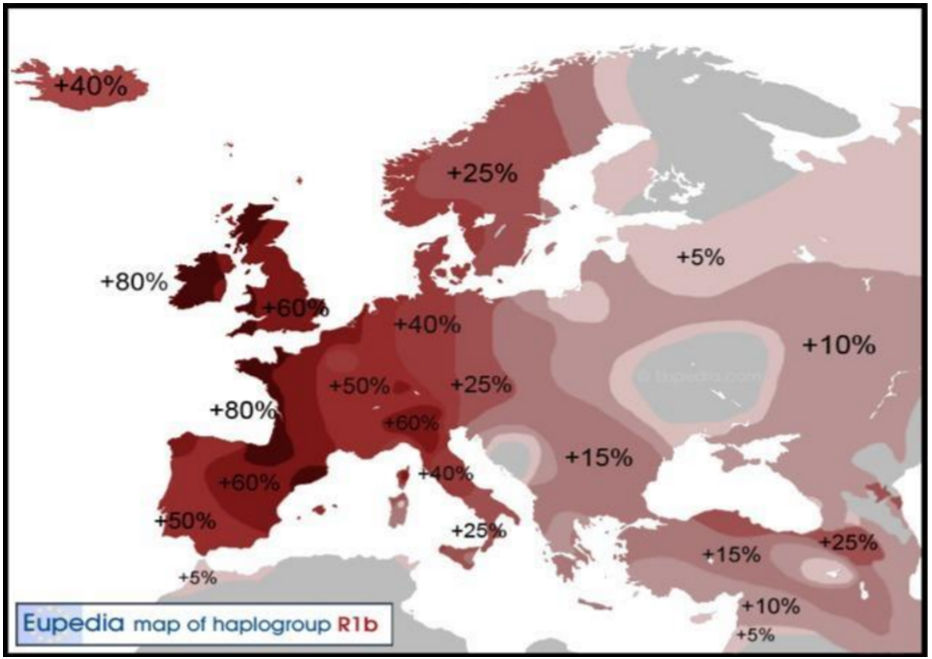 Gráfico 2. Distribución del haplogrupo R1b en Europa (Gómez Moreno, 2020, 16).
Gráfico 2. Distribución del haplogrupo R1b en Europa (Gómez Moreno, 2020, 16).
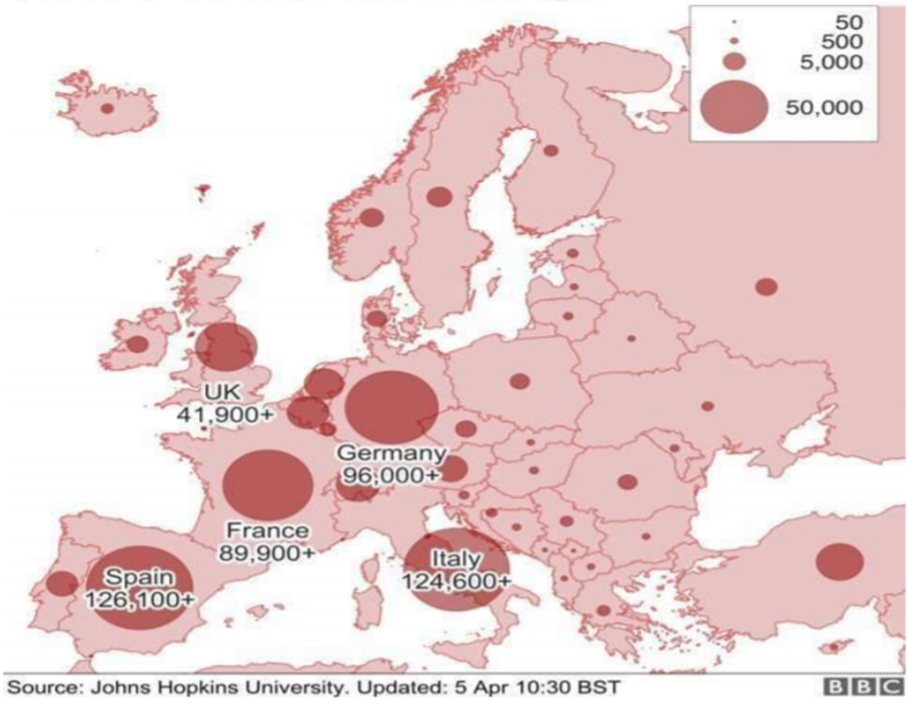 Gráfica 3. Incidencia del COVID-19 en Europa, en 05/04/2020 ((Gómez Moreno, 2020, 26)
Gráfica 3. Incidencia del COVID-19 en Europa, en 05/04/2020 ((Gómez Moreno, 2020, 26)
Recordemos que un haplotipo es la combinación de varios genes de loci estrechamente ligados sobre un mismo cromosoma que tienden a transmitirse como una unidad hereditaria de padres a hijos por la baja probabilidad de recombinación. Obviamente, en el caso del cromosoma Y dependería que los loci estuvieran localizados en el segmento diferencial o apareante en relación con el cromosoma X. Se incluye un gráfico donde se muestra la frecuencia de distribución del haplotipo R1b en Europa continental, regiones mediterráneas, Próximo Oriente, África, etc. (gráfico 4).
Gráfico 4. Mapa de distribución del haplogrupo R1b en Europa, África y Oriente (https://www.eupedia.com/europe/Haplogroup_R1b_Y-DNA.shtml)
¿Por qué hay una relación entre el mencionado haplotipo y la distribución de la pandemia del coronavirus? Yo no he encontrado explicación alguna en la literatura.
Aunque el estudio del profesor Gómez Moreno se refiere especialmente a Europa, también el continente americano se ha visto afectado por la pandemia en la misma medida que en Europa. Ello es lógico porque el haplotipo R1b pudo llegar a los Estados Unidos por su ascendencia inglesa, escocesa e irlandesa que comparten altas frecuencias del mismo. Por otro lado, los países latinoamericanos (Argentina, Chile, Méjico, etc.) tienen la ascendencia española e italiana y Brasil portuguesa.
En relación con África (gráfico 3), afortunadamente, la escasa frecuencia con la que se presenta el haplotipo R1b puede atenuar la pandemia en muchos de sus países cuyos recursos sanitarios son precarios.
Bibliografía
Cavalli-Sforza, L.L. Genes, Peoples and Languages. Scientific American (1991), 265(5), 104–111.
Cavalli-Sforza, L.L.; A. Piazza; P. Menozzi; J. Mountain. Reconstruction of human evolution: bringing together genetic, archaeological, and linguistic data. Proc. Nat. Acad. Sci. (1988), 85(16), 6002-6006 [doi.org/10.1073/pnas.85.16.6002].
Gómez Moreno, Á. Coronavirus, Genética de las poblaciones y Humanidades. [Lección de Clausura del Curso Académico 2019-20 en el seno del Institut Superior de Investigació Cooperativa IVITRA [ISIC-IVITRA] de la Universitat d’Alacant, en el marco del 15è Simposi Internacional en Noves Tendències en I+D+i en Literatura, Llengua, Ensenyament i TIC]. In Antonio Cortijo, Vicent Martines, Armando Alexandre dos Santos (org.) Mirabilia (2020), 30(1): https://www.revistamirabilia.com/sites/default/files/pdfs/01._gomezmoreno.pdf [consulta: 23/05(2020].
Sutherland, W. Parallel extinction risk and global distribution of languages and species. Nature (2003), 423, 276–279 [doi.org/10.1038/nature01607].
Venter, J.C. A Life Decoded: My Genome: My Life. New York, The Viking Press, 2007. [traducción al español, por Jesús Fabregat, Espasa-Calpe, Madrid, 2008].
Juan-Ramón Lacadena
Académico de la RANF

